정보
-
업무명 : R을 이용한 부트스트랩 방법을 이용한 표본 평균 및 히스토그램 가시화
-
작성자 : 이상호
-
작성일 : 2020-10-18
-
설 명 :
-
수정이력 :
내용
[개요]
-
안녕하세요? 웹 개발 및 연구 개발을 담당하고 있는 해솔입니다.
-
다년간 축적된 경험 (기상학 학술 보고서 및 국/영문 학술 논문 게재, 블로그 운영, IT 회사 웹 개발 담당) 및 노하우를 바탕으로 개개인에게 맞춤형 솔루션을 수행할 수 있습니다.
-
특히 재능 플랫폼 (크몽, 오투잡, 해피캠퍼스, 레포트 월드)에서 누구보다도 경쟁력 있는 가격으로 양질의 서비스를 제공하고 있습니다.
-
아스키 형식의 텍스트 (text) 파일부터 과학자료 형식 (HDF, H5, NetCDF, Grib, Grb) 및 Data Base (DB) 자료까지 다양한 형태의 자료를 이용하여 수집, 전처리, 분석, 시각화해 드립니다.
-
또한 웹 사이트에 대한 정보를 이용한 웹 크롤링 및 그에 따른 엑셀 및 DB 구축도 가능합니다.
-
아울러 기초 통계 (빈도분포, Prired t-test, Wilcoxn 등)에서 지도/비지도 학습을 통한 회귀모형 구축에 이르기 까지 효율적인 통계 정보를 제공합니다.
-
최근 대한민국의 후속위성인 천리안위성 2A호 웹 서비스 서브시스템 및 환경위성 2B호 통합 자료처리 서브시스템에 대한 웹 개발을 수행하였습니다.
-
-
그리고 해솔 블로그에서는 다양한 기상학/천문학 정보와 더불어 사무 자동화/프로그래밍 언어를 소개하오니 방문 부탁드립니다.
-
좋은 하루 보내세요.
[재능플랫폼] 오투잡
[IT개발 - 응용프로그래밍] 통계 분석, 데이터 분석, 시각화를 성실하게 해 드립니다. - 재능마켓 �
판매가격:10,000원, [소개] - 데이터산업진흥원 데이터 가공 공급기업 선정 - 정보통신산업 진흥원 데이터 가공 공급기업 선정 - 다년간 축적된 경험 노하우를 바탕으로 개개인에게 맞춤형 솔루션�
www.otwojob.com
[재능플랫폼] 크몽
데이터수집, 파싱, 크롤링 해 드립니다. | 50,000원부터 시작 가능한 총 평점 0점의 IT·프로그래밍,
0개 총 작업 개수 완료한 총 평점 0점인 shlee1990의 IT·프로그래밍, 데이터분석·리포트, 데이터 마이닝·크롤링 서비스를 0개의 리뷰와 함께 확인해 보세요. IT·프로그래밍, 데이터분석·리포트, 데
kmong.com
요청
[세부 사항]
-
diabetes 데이터에서 나이가 많은 그룹이 나이가 작은 그룹에 비하여 logCpeptide의 평균이 크다는 가설을 임의화 방법으로 테스트하고자 한다. p-값을 산출하고 영가설 하에서 확률적으로 동등한 표본 평균 간 차이들의 히스토그램에 관측된 차이를 marking하라
-
앞 문제의 계속. 나이가 많은구룹의 평균 logCpeptide와 나이가 작은 그룹의 평균 logCpeptide 간 차이에 대한 신뢰구간을 붓스트랩 방법으로 산출하고자 한다. 95% 신뢰구간을 산출하고 붓스트랩 관측 차이들의 히스토그램에 marking하라
완료
[사용 OS]
-
Windows 10
[사용 언어]
-
R v4.0.2
[명세]
문제 1
-
라이브러리 및 자료 읽기
library(bootstrap)
data(diabetes)
-
age > 10을 기준으로 인덱스 설정
-
해당 인덱스의 여부에 따라 그룹 설정 (groupA, groupB)
-
각 그룹에 대한 평균 (meanGroupA, meanGroupB) 및 차이 (r)
# 모집단
ind = which(diabetes$age > 10)
groupA = diabetes[ind, ]
groupB = diabetes[-ind, ]
meanGroupA = mean(groupA$logCpeptide, na.rm = TRUE)
meanGroupB = mean(groupB$logCpeptide, na.rm = TRUE)
r = meanGroupA - meanGroupB
r
-
표본 평균의 차이 (r)를 모집단으로 가정하고 무작위 추출 (sample)을 통해 부트스트랩 수행
-
무작위 추출 결과를 이용하여 히스토그램
-
P값 계산
n.repeat = 1000
r.random = rep(0, n.repeat)
count = 0
for (k in 1:n.repeat) {
sampleList = base::sample(1:nrow(diabetes), replace = TRUE)
# sample(X, N, replace=F)
r.data = diabetes[sampleList, ]
r.ind = which(r.data$age > 10)
r.groupA = r.data[r.ind, ]
# r.groupB = diabetes[-r.ind, ]
r.meanGroupA= mean(r.groupA$logCpeptide, na.rm = TRUE)
r.star = r.meanGroupA - meanGroupB
r.random[k] = r.star
if (r.star <= r) count = count + 1
}
hist(r.random, nclass=20)
text(r, 0, "|", col = "red", cex = 2.0)
p.value = count/n.repeat
p.value

문제 2
-
age > 10을 기준으로 인덱스 설정
-
해당 인덱스의 여부에 따라 그룹 설정 (r.groupA, r.groupB)
-
각 그룹에 대한 평균 (r.meanGroupA, r.meanGroupB) 및 차이 (r.star)
-
표본 평균의 차이 (r)를 모집단으로 가정하고 무작위 추출 (sample)을 통해 부트스트랩 수행
n.repeat = 1000
r.boot = rep(0, n.repeat)
count = 0
for (k in 1:n.repeat) {
sampleList = base::sample(1:nrow(diabetes), replace = FALSE)
r.ind = which(diabetes[sampleList, ]$age > 10)
r.groupA = diabetes[r.ind, ]
r.groupB = diabetes[-r.ind, ]
r.meanGroupA= mean(r.groupA$logCpeptide, na.rm = TRUE)
r.meanGroupB= mean(r.groupB$logCpeptide, na.rm = TRUE)
r.star = r.meanGroupA - r.meanGroupB
r.boot[k] = r.star
}
-
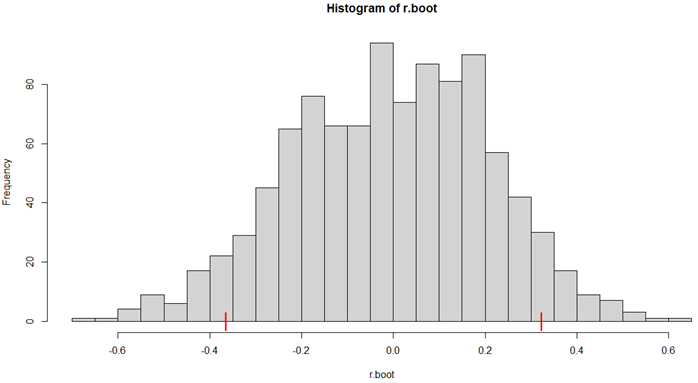
무작위 추출 결과 (r.boot)를 이용하여 히스트그램
-
quantile를 통해 신뢰구간 90% 및 95% 계산
hist(r.boot, nclass=20)
# 신뢰구간 90%
conf.1 = quantile(r.boot, p = 0.05)
conf.2 = quantile(r.boot, p = 0.95)
# 신뢰구간 95%
conf.1 = quantile(r.boot, p = 0.025)
conf.2 = quantile(r.boot, p = 0.975)
text(conf.1, 0, "|", col = "red", cex = 2.0)
text(conf.2, 0, "|", col = "red", cex = 2.0)
round(c(conf.1, conf.2), 2)

[소스 코드]
#===============================================================================================
# Routine : Main R program
#
# Purpose : 재능상품 오투잡
#
# Author : 해솔
#
# Revisions: V1.0 May 28, 2020 First release (MS. 해솔)
#===============================================================================================
#=========================================
# 문제 1
#=========================================
# 1. diabetes 데이터에서 나이가 많은 그룹이 나이가 작은 그룹에 비하여 logCpeptide의 평균이 크다는 가설을 임의화 방법으로 테스트하고자 한다.
# p-값을 산출하고 영가설 하에서 확률적으로 동등한 표본 평균 간 차이들의 히스토그램에 관측된 차이를 marking하라.
library(bootstrap)
data(diabetes)
# 모집단
ind = which(diabetes$age > 10)
groupA = diabetes[ind, ]
groupB = diabetes[-ind, ]
meanGroupA = mean(groupA$logCpeptide, na.rm = TRUE)
meanGroupB = mean(groupB$logCpeptide, na.rm = TRUE)
r = meanGroupA - meanGroupB
r
# 표본 평균
n.repeat = 1000
r.random = rep(0, n.repeat)
count = 0
for (k in 1:n.repeat) {
sampleList = base::sample(1:nrow(diabetes), replace = TRUE)
# sample(X, N, replace=F)
r.data = diabetes[sampleList, ]
r.ind = which(r.data$age > 10)
r.groupA = r.data[r.ind, ]
# r.groupB = diabetes[-r.ind, ]
r.meanGroupA= mean(r.groupA$logCpeptide, na.rm = TRUE)
r.star = r.meanGroupA - meanGroupB
r.random[k] = r.star
if (r.star <= r) count = count + 1
}
hist(r.random, nclass=20)
text(r, 0, "|", col = "red", cex = 2.0)
p.value = count/n.repeat
p.value
#=========================================
# 문제 2
#=========================================
# 앞 문제의 계속. 나이가 많은구룹의 평균 logCpeptide와 나이가 작은 그룹의 평균 logCpeptide 간 차이에 대한 신뢰구간을 붓스트랩 방법으로 산출하고자 한다.
# 95% 신뢰구간을 산출하고 붓스트랩 관측 차이들의 히스토그램에 marking하라.
n.repeat = 1000
r.boot = rep(0, n.repeat)
count = 0
for (k in 1:n.repeat) {
sampleList = base::sample(1:nrow(diabetes), replace = FALSE)
r.ind = which(diabetes[sampleList, ]$age > 10)
r.groupA = diabetes[r.ind, ]
r.groupB = diabetes[-r.ind, ]
r.meanGroupA= mean(r.groupA$logCpeptide, na.rm = TRUE)
r.meanGroupB= mean(r.groupB$logCpeptide, na.rm = TRUE)
r.star = r.meanGroupA - r.meanGroupB
r.boot[k] = r.star
}
hist(r.boot, nclass=20)
conf.1 = quantile(r.boot, p = 0.05)
conf.2 = quantile(r.boot, p = 0.95)
text(conf.1, 0, "|", col = "red", cex = 2.0)
text(conf.2, 0, "|", col = "red", cex = 2.0)
round(c(conf.1, conf.2), 2)
[결과물]
- 결과 보고서



참고 문헌
[논문]
- 없음
[보고서]
- 없음
[URL]
- 없음
문의사항
[기상학/프로그래밍 언어]
- sangho.lee.1990@gmail.com
[해양학/천문학/빅데이터]
- saimang0804@gmail.com
'자기계발 > 재능상품' 카테고리의 다른 글
| [재능상품] R을 이용한 학술연구정보서비스 크롤링 및 클라우드 시각화 (0) | 2020.11.10 |
|---|---|
| [재능상품] Python을 이용한 CLT (Central Limit Theorem) 시연 (0) | 2020.10.18 |
| [재능상품] R을 이용한 프로그래밍 과제 (0) | 2020.10.18 |
| [재능상품] Shell Script를 이용한 Bingo 함수 버전, 재귀 처리, 숫자 야구 (0) | 2020.10.18 |
| [재능상품] R을 이용한 점수 변화 그래프 및 바람 장미 시각화 (0) | 2020.10.12 |

