정보
-
업무명 : 아이디엘 HDF 형식인 Terra/MODIS 극궤도 기상 위성 자료를 이용한 총 오존량 가시화
-
작성자 : 이상호
-
작성일 : 2019-08-29
-
설 명 :
-
수정이력 :
-
2020-03-24 : 소스 코드 명세 추가
-
내용
[개요]
-
안녕하세요? 기상 연구 및 웹 개발을 담당하고 있는 해솔입니다.
-
TOMS (Total Ozone Mapping Spectrometer)는 1978 년 11 월 NIMBUS 위성 7호에서 처음 시작하여 매일 총 오존량의 전 세계 분포를 측정했습니다.
-
이러한 TOMS는 1993년 05월 06일까지 약 14.5 년 동안 계속 관측하였으며 남극의 봄 기간에 심한 오존층 파괴로 인한 오존 구멍이 발견되었을 때 TOMS의 이름이 널리 알려졌습니다.
-
TOMS 데이터는 남극 대륙의 오존 구멍 면적의 범위뿐만 아니라 80년대 초 이후의 시간에 따른 오존 구멍의 발달을 분명히 보여주었습니다.
-
그 밖에 Meteor 위성 3호, ADEOS 위성들도 우주에서 총 오존 관측을 수행했으며 최근에 많은 극궤도 기상위성들이 오존층 모니터링을 하고 있습니다.
-
오늘 포스팅에서는 HDF 형식인 Terra/MODIS 극궤도 기상 위성 자료를 이용한 총 오존량 가시화를 소개해 드리고자 합니다.
[특징]
-
HDF 기상 위성 자료를 이용하여 정성적으로 가시화하기 위한 도구가 필요하며 이 프로그램은 이러한 목적을 달성하기 위해 고안된 소프트웨어
[기능]
-
Terra/MODIS 극궤도 기상 위성 자료에 대한 위경도 및 총 오존량 읽기
-
위경도 및 총 오존량에 대한 통계 수치 (최대값, 최소값, 평균) 확인
-
지상 관측소의 위/경도를 이용하여 맵핑
-
도법 (cylindrical, satellite)에 따라 가시화 및 컬러바 설정
-
이미지 저장
[사용법]
-
입력 자료를 동일 디렉터리에 위치
-
소스 코드를 실행 (idl -e Visualization_Using_Terra_MODIS_Data)
-
가시화 결과를 확인
[사용 OS]
-
Linux
-
Windows 10
[사용 언어]
-
IDL v8.5
소스 코드
-
해당 작업을 하기 위한 컴파일 및 실행 코드는 IDL로 작성되었으며 가시화를 위한 라이브러리는 Coyote's Guide to IDL Programming를 이용하였습니다.
-
소스 코드는 단계별로 수행하며 상세 설명은 다음과 같습니다.
-
1 단계는 주 프로그램은 작업 경로 설정, HDF 파일 읽기, 변수 전처리하여 메모리상에 저장하고 가시화를 위한 초기 설정합니다.
-
2 단계는 plot 또는 contour 매핑에 따라 영상 장면 표출하여 이미지 형식으로 저장합니다. 이 과정에서 포스트 스크립트 (PS) 형식에서 PNG로 변환합니다.
-
[명세]
-
작업 환경 설정
cd, 'D:\할 일\Program\각종 자료\Aerosol mapping+RGB\modis hdf_ps_RGB x\Ozone'
-
파일 찾기
hdf_files=file_search('./INPUT/MOD07_L2*.hdf',count=count)
-
HDF 파일 읽기
-
반복문 수행하면서 hdf_files 및 hdf_sd_start 파일 읽기
-
for k=0,count-1, 1 do begin
hdf_file=hdf_files(k)
print, hdf_file
sd_id=hdf_sd_start(hdf_file, /read)
# 생략
endfor
-
각 변수에 대한 전처리
-
hdf_sd_nametoindex 및 hdf_sd_getdata를 통해 위도 (lat), 경도 (lon), 총 오존량 (var) 디지털값 설정
-
hdf_sd_attrfind를 통해 결측값 (_FillValue), 유효 범위 (valid_range), scale_factor, add_offset 변수 설정
-
where를 통해 결측값 및 유효범위에 제외한 후 scale_factor 및 add_offset를 통해 디지털값을 물리적 변수로 변환
-
sds_index = hdf_sd_nametoindex(sd_id, 'Latitude')
sds_id = hdf_sd_select(sd_id, sds_index)
sds_id_fill = hdf_sd_attrfind(sds_id, '_FillValue')
hdf_sd_attrinfo, sds_id, sds_id_fill, data=fill
sds_id_valid = hdf_sd_attrfind(sds_id, 'valid_range')
hdf_sd_attrinfo, sds_id, sds_id_valid, data=valid
sds_id_scale = hdf_sd_attrfind(sds_id, 'scale_factor')
hdf_sd_attrinfo, sds_id, sds_id_scale, data=scale
sds_id_add = hdf_sd_attrfind(sds_id, 'add_offset')
hdf_sd_attrinfo, sds_id, sds_id_add, data=offset
hdf_sd_getdata, sds_id, data
data=float(data)
loc1 = where( ((data lt valid[0]) or (data gt valid[1]) or (data eq fill[0])), nloc1, complement=loc2, ncomplement=nloc2 )
if (nloc1 gt 0) then data[loc1] = !values.f_nan
if (nloc2 gt 0) then data[loc2] = scale[0]*(data[loc2]-offset[0])
lat = data
sds_index = hdf_sd_nametoindex(sd_id, 'Longitude')
sds_id = hdf_sd_select(sd_id, sds_index)
sds_id_fill = hdf_sd_attrfind(sds_id, '_FillValue')
hdf_sd_attrinfo, sds_id, sds_id_fill, data=fill
sds_id_valid = hdf_sd_attrfind(sds_id, 'valid_range')
hdf_sd_attrinfo, sds_id, sds_id_valid, data=valid
sds_id_scale = hdf_sd_attrfind(sds_id, 'scale_factor')
hdf_sd_attrinfo, sds_id, sds_id_scale, data=scale
sds_id_add = hdf_sd_attrfind(sds_id, 'add_offset')
hdf_sd_attrinfo, sds_id, sds_id_add, data=offset
hdf_sd_getdata, sds_id, data
data = float(data)
loc1 = where( ((data lt valid[0]) or (data gt valid[1]) or (data eq fill[0])), nloc1, complement=loc2, ncomplement=nloc2 )
if (nloc1 gt 0) then data[loc1] = !values.f_nan
if (nloc2 gt 0) then data[loc2] = scale[0]*(data[loc2]-offset[0])
lon = data
sds_index = hdf_sd_nametoindex(sd_id, 'Total_Ozone')
sds_id = hdf_sd_select(sd_id, sds_index)
sds_id_fill = hdf_sd_attrfind(sds_id, '_FillValue')
hdf_sd_attrinfo, sds_id, sds_id_fill, data=fill
sds_id_valid = hdf_sd_attrfind(sds_id, 'valid_range')
hdf_sd_attrinfo, sds_id, sds_id_valid, data=valid
sds_id_scale = hdf_sd_attrfind(sds_id, 'scale_factor')
hdf_sd_attrinfo, sds_id, sds_id_scale, data=scale
sds_id_add = hdf_sd_attrfind(sds_id, 'add_offset')
hdf_sd_attrinfo, sds_id, sds_id_add, data=offset
hdf_sd_getdata, sds_id, data
data=float(data)
loc1 = where( ((data lt valid[0]) or (data gt valid[1]) or (data eq fill[0])), nloc1, complement=loc2, ncomplement=nloc2 )
if (nloc1 gt 0) then data[loc1] = !values.f_nan
if (nloc2 gt 0) then data[loc2] = scale[0]*(data[loc2]-offset[0])
var = data
hdf_sd_endaccess, sds_id
hdf_sd_end, sd_id
-
위/경도 및 오존량에 대한 통계 수치 확인
-
최소값 : min
-
평균 : mean
-
최대값 : max
-
;============================
; Statistical analysis
;============================
print, min(lon, /nan), mean(lon, /nan), max(lon, /nan)
print, min(lat, /nan), mean(lat, /nan), max(lat, /nan)
print, min(var, /nan), mean(var, /nan), max(var, /nan)
-
기타 설정
-
표출 영역 (latmin, lonmin, latmax, lonmax) 설정
-
지상 관측소의 위도 (point_lat), 경도 (point_lon), 지점명 (point_name) 설정
-
;==================================
; Option
;==================================
start_color=0 & end_color=255 & colorn=end_color-start_color+1
latmin=-90 & lonmin=0 & latmax=90 & lonmax=360 ; satellite
point_lon = [126.14, 126.16, 126.93]
point_lat = [37.23, 33.29, 37.56]
point_name = ['a', 'b', 'c']
-
가시화를 위한 초기 설정
-
set_plot를 통해 포스트 스크립트 (Post Script, PS) 형식으로 저장 및 세부 옵션
-
그림 저장명 : filename
-
폰트 : /SCHOOLBOOK
-
폰트 굵게 : /BOLD
-
-
usersym를 통해 매핑을 위한 심볼 설정
-
중심 위도 (clat), 중심 경도 (clon) 설정
-
cgmap_set를 통해 도법 (satellite) 설정
-
n_color = 256
ps_name = file_basename(hdf_file, '.hdf') ; file name Change
print, ps_name
set_plot,'ps'
device, filename=ps_name+'.ps', decomposed=0, bits=8, /color, xsize=13, ysize=12, /inches, $
/SCHOOLBOOK, /BOLD
!p.font=0 & !p.charsize=2.0 & !p.charthick=1.6 & !p.multi=[0,1,1]
usersym, [0.1,0.1,-0.1,-0.1,0.1], [-0.1,0.1,0.1,-0.1,-0.1], /fill
clat = 0.0 & clon = 128.2
cgmap_set, /satellite, sat_p=[42159.934825,0,0], clat, clon, charsize=1.0, limit=[-90,-180,90,180], $
position=[0.10,0.15,0.9,0.95], /isotropic, color='white'
zmin = 300. & zmax = 500.
cgloadct, 33, bottom=0, ncolor=n_color
-
매핑을 위한 설정 및 가시화 (1) : points
-
총 오존량의 최대/최소값 : zmin 및 zmax
-
cgloadct를 통해 컬러바 설정
-
위/경도 개수 (dims)만큼 반복문을 수행하면서 분기문 수행
-
총 오존량 > 0 이상인 경우
-
최대/최소 위/경도인 경우
-
-
plots를 통해 위/경도에 따른 총 오존량 컬러 (color) 가시화
-
;===========================
; Mapping : plot
;===========================
zmin = 300. & zmax = 500.
cgloadct, 33, bottom=0, ncolor=n_color
tvlct, r, g, b, /Get
latmin=32 & lonmin=120 & latmax=42 & lonmax=132
for i=0L, dims[0]-1 do begin
for j=0L, dims[1]-1 do begin
val=var[i,j]
if(val gt 0 ) then begin
if( (lat[i,j] gt latmin) and (lat[i,j] lt latmax) ) then begin
if( (lon[i,j] gt lonmin) and (lon[i,j] lt lonmax) ) then begin
plots, lon[i,j], lat[i,j], psym=8, symsize=2.5, color=start_color+colorn*(val-zmin)/(zmax-zmin)
endif
endif
endif
endfor
endfor
-
매핑을 위한 설정 및 가시화 (2) : contour
-
총 오존량의 최대/최소값 : zmin 및 zmax
-
cgloadct를 통해 컬러바 설정
-
cgcontour를 통해 총 오존량 등고선 가시화
-
;========================================
; Mapping : Contour fill, contour
;========================================
zmin = 300. & zmax = 500.
levels=255
step=(zmax-zmin)/levels
userlevels=findgen(levels)*step + zmin
cgloadct, 33, ncolors=levels, bottom=0
cgcontour, var, lon, lat, /overplot, /cell_fill, nlevels=levels, $
C_THICK=3, C_CHARSIZE=2, C_CHARTHICK=2, c_color=indgen(levels), levels=userlevels
-
도법에 대한 격자 보조선 설정
-
cgmap_grid를 기준 위/경도 (lats, lons)에 따라 텍스트 (lats_names, lons_names) 및 격자 보조선 삽입
-
cgtext를 통해 그림 제목 삽입
-
lats = [-90, -60, -30, 0, 30, 60, 90]
lats_names = ['',textoidl('60\circS', font=0), textoidl('30\circS', font=0), ' ', textoidl('30\circN', font=0),textoidl('60\circN', font=0), '']
lons = [10, 70, 100, 130, 160, -170, -10]
lons_names = ['',textoidl('70\circE'), textoidl('100\circE'), textoidl('130\circE'), textoidl('160\circE'), textoidl('170\circW'),'']
cgmap_grid, color='black', /label, lats=lats, latlabel=160, lonlabel=-5, latnames=lats_names, lons=lons, lonnames=lons_names, $
clip_text=0, linestyle=1, noclip=0, /horizon, charsize=1.7
cgtext, 0.5, 0.965, ps_name, charsize=2, charthick=1.2, alignment=0.5, color='black', font=0, /normal ; GOCI(1)
-
포스트 스크립트 형식 (PS)을 PNG로 변환
-
device를 통해 포스크 스크립트 정상 종료
-
convert를 통해 PNG 이미지 형태로 변환
-
device, /close_file
com = 'convert -flatten -background white '+ps_name+'.ps'+' '+'OUTPUT/'+file_basename(ps_name, '.ps')+'.png'
spawn, com
file_delete, ps_name+'.ps'
-
MODIS 자료를 이용하여 한반도 총 오존량 분포
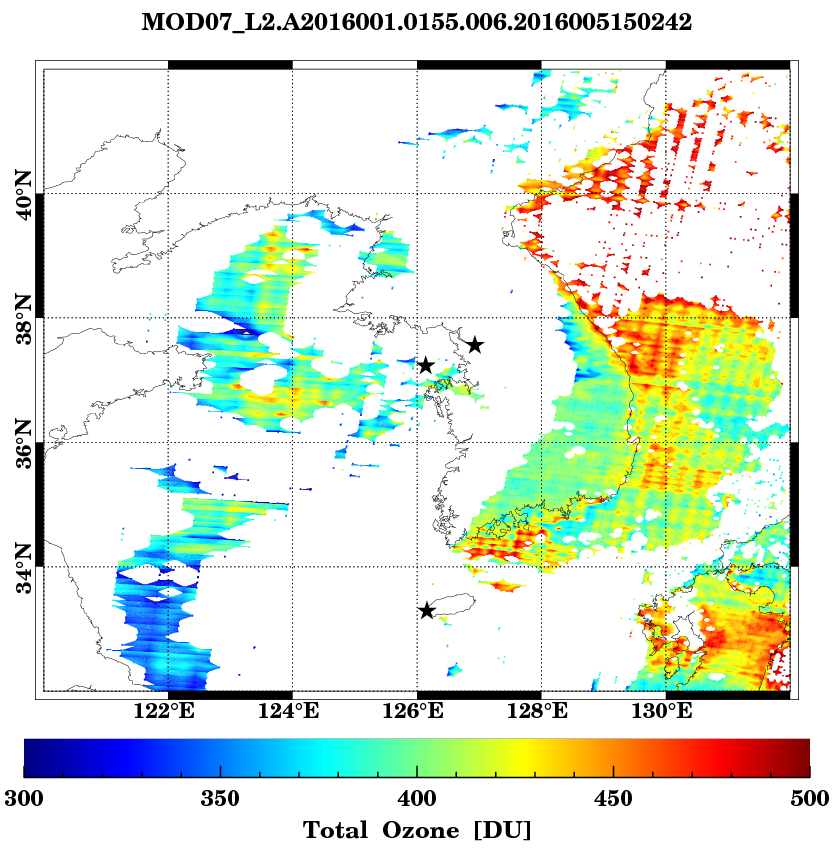
-
MODIS 자료를 이용하여 동아시아 총 오존량 분포
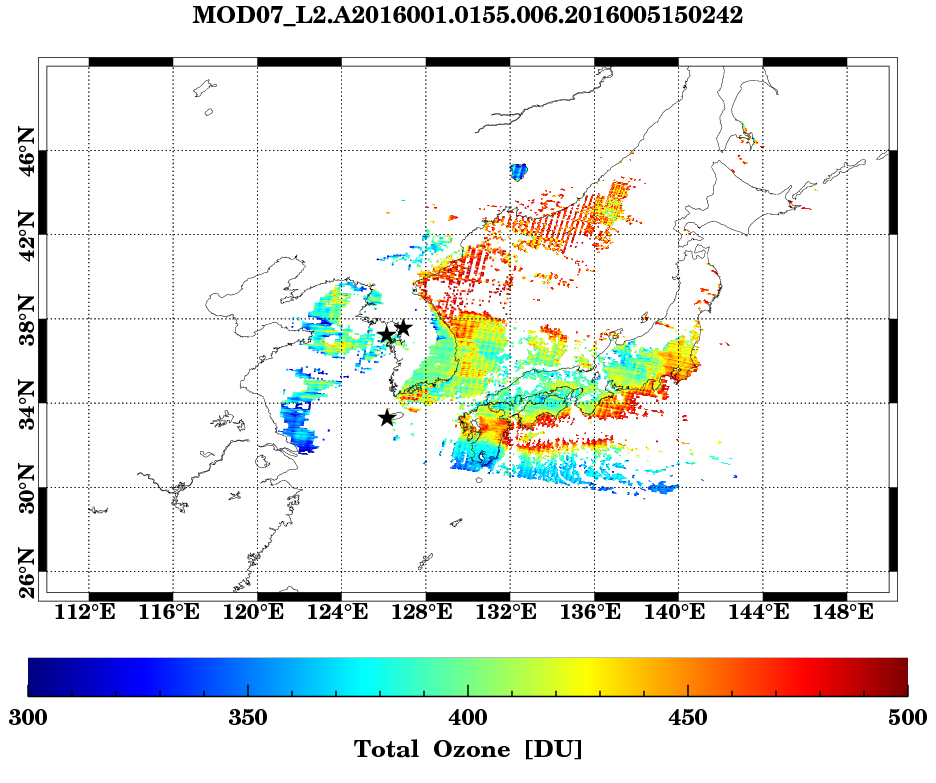
-
MODIS 자료를 이용하여 전구 총 오존량 분포
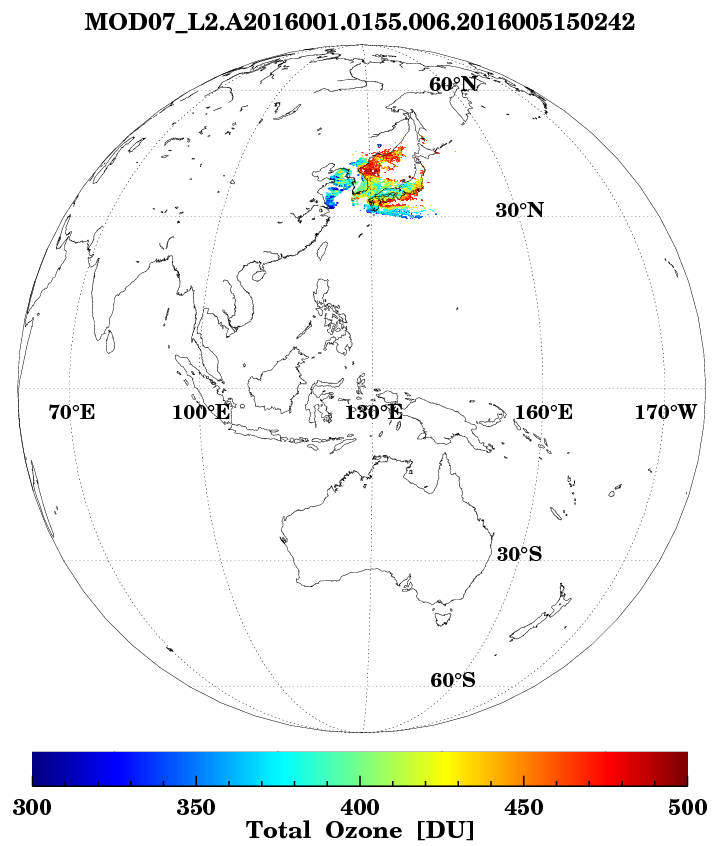
[전체]
참고 문헌
[논문]
- 없음
[보고서]
- 없음
[URL]
- 없음
문의사항
[기상학/프로그래밍 언어]
- sangho.lee.1990@gmail.com
[해양학/천문학/빅데이터]
- saimang0804@gmail.com

